|
Veterinaria Trop. 26(1):7-25. 1995. CARACTERIZACIÓN MOLECULAR DE CEPAS DEL VIRUS DE LA BRONQUITIS INFECCIOSA AVIAR AISLADAS EN LA REGIÓN CENTRAL DE VENEZUELA Mariela Brett*
, Carmen de Noguera** , Carlos Ouiroz** ,
Morela de Rolo**, *Profesional en actividad
privada. Facultad de Ciencias Veterinarias. Recibido: agosto 30, 2001. |
||||
|
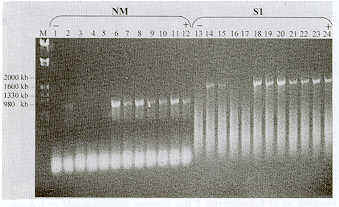
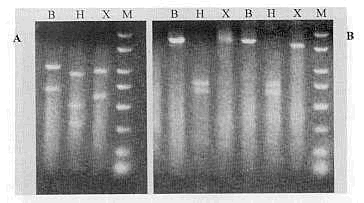
RESUMEN Se realizó un estudio con el fin de caracterizar genómicamente 9 cepas del virus de la bronquitis infecciosa aviar (BIA), las cuales fueron aisladas de pollos de engorde procedentes de granjas en la región central del país. La técnica utilizada fue la Transcripción Reversa- Reacción en cadena de la Polimerasa-Polimorfismo de la Longitud de los Fragmentos de Restricción (RT -PCR- RFLP). De las 9 cepas, 7 correspondieron al serotipo Massachusetts y 2 a un serotipo desconocido, que no coincide con los patrones estándar de restricción, identificada como "variante genotípica". Cabe señalar que el serotipo Massachusetts que había sido aislado en el país hasta el momento. La presencia de serotipos desconocidos pudieran surgir como consecuencia dc prácticas inadecuadas de manejo, de ahí la importancia de la identificación molecular de las cepas de campo a fin de establecer planes de prevención y control adecuados. Los resultados obtenidos demuestran la alta sensibilidad y especificidad de la prueba RT-PCR-RFLP en el diagnóstico de la BIA. Palabras Claves: Bronquitis infecciosa; variante genotípica; serotipo; RT-PCR-RFLP. INTRODUCCIÓN La bronquitis infecciosa aviar (BIA) es una enfermedad causada por un coronavirus que afecta el sistema respiratorio y urogenital de los pollos de engorde, ponedoras y reproductoras, induciendo grandes pérdidas económicas a la industria avícola, debido principalmente al bajo rendimiento de los pollos de engorde, disminución en la producción y calidad de los huevos, así como de la fertilidad (Crinion et al.,1970; Munner et al.,1987). La BIA es controlada mediante el uso de vacunas vivas e inactivadas, pero la amplia variabilidad antigénica que posee el virus hace difícil un control eficaz (King y Cavanagh, 1991; Picault et al, 1986), presentándose brotes de la enfermedad en parvadas totalmente vacunadas, debido probablemente a que los aislados de campo difieren antigénicamente de los serotipos vacunales (Gelb et al., 1991), por lo cual la BIA continua siendo un problema en la cría de las aves comerciales al no existir protección cruzada entre los diferentes serotipos o variantes del virus (Jackwood, 1999). A nivel mundial con frecuencia se aíslan serotipos variantes a partir de gallinas ponedoras criadas en complejos avícolas de múltiples edades y con alta densidad (Gelb et al., 1997), en consecuencia, granjas de pollos de engorde cercanas a esas granjas pueden fácilmente contaminarse con estos serotipos (Gelb et al., 1991), por lo cual se hace necesario el aislamiento y caracterización de las cepas en cada uno de los brotes. Tradicionalmente la BIA es diagnosticada por aislamiento e identificación del virus con pruebas convencionales, la neutralización viral (NV) es la prueba definitiva para determinar el serotipo del virus de la BIA, pero esta es una prueba muy laboriosa y consume mucho tiempo. Un nuevo y rápido método basado en la biología molecular ha sido diseñado para la identificación genómica de este virus llamado RT-PCR-RFLP, con el que se analiza el ácido nucleico, y pueden diferenciarse serotipos conocidos incluyendo cepas variantes (Jackwood y Jackwood, 1998; King y Cavanagh, 1991). Este método ha sido reconocido por su alta sensibilidad y especificidad en el diagnóstico del virus de la BIA (Jackwood et al.,1992). MATERIALES Y METODOS Cepas de virus. 9 cepas del virus de la BIA fueron aisladas de pollos de engorde provenientes de 30 granjas ubicadas en los estados Aragua, Carabobo, Cojedes y Miranda. Las aves presentaban signos clínicos sospechosos a la enfermedad. Para el aislamiento se inocularon huevos embrionados susceptibles, de 9 a 11 días de edad, libres de anticuerpos específicos contra la BIA, siguiendo la técnica descrita por Gelb y Jackwood (1998). Los fluidos alantoideos (FA) sirvieron como fuente de virus, los cuales se mantuvieron en congelación hasta su caracterización. Extracción del ARN. Fue obtenido a partir de FA, utilizando la técnica de extracción con TRIzol Ls (GIBCO BRL, NY), usada por otros investigadores (Wang et al., 1999), modificado al agregar 5 µl de Polyacryl Carrier, con el fin de purificar el ARN, el cual fue restituido en 50 µ1 de agua tratada con Dietil Pirocarbonato y mantenido en congelación a -30 °C. Caracterización molecular del virus de BIA. La caracterización fue realizada siguiendo el procedimiento descrito por Kwon et al. (1993). Los primeros utilizados con la secuencia para el gen que codifica la proteína N fueron: NIBVPCR. 5' TGG CAC CGC TAC CTT CAA ACT TGG GCG G 3'. Para el gen que codifica la proteína M: MIBVPCR 5' TAA GTC TTC AGT GGC TTG CTA AGT GTG AAC C 3'. Para el gen que codifica la subunidad S1 de la proteína S: S1OLIGO 5': TGA AAC TGA ACA AAA GAC y S1OLIGO3: CAT AAC TAA CAT AAG GGC AA; todos fueron diseñados y sintetizados en la Universidad de Georgia (Molecular Genetics Facility) USA, a una concentración final de 0,5 ng/ml. La RT-PCR se realizó utilizando el kit TitanR (One Tube RT-PCR System, Boehringer Mannheim) y fue conducido en un termo-ciclador Gene Amp PCR System 9 600 y 2 400 de Perkin Elmer, con el fin de amplificar los genes que codifican las proteínas estructurales del virus de la BIA (N/M y S1). A cada una de las muestras se le realizó dos RT -PCR por separado, una para el gen de la proteína N/M la cual identifica al virus de la BIA y otra para el gen de la proteína S1 la cual por efecto de las enzimas de la restricción (RFLP) permite la identificación del serotipo. La trascripción reversa se realizó por una hora a 50 °C. La amplificación fue realizada en 35 ciclos continuos de tres fases:. desnaturalización a 94 °C x 30 s, la unión de los primers a 55 °C x 30 s, y polimerización a 68 °C x 1,5 min. El producto de esta amplificación fue analizado por electroforesis y observado en un transiluminador de luz ultravioleta. Para el RFLP se utilizó el producto amplificado por PCR del gen de la subunidad S1 de la proteína S, el cual fue sometido a digestión con tres enzimas de restricción: BestY -I, Hae-III y Xcm-I. El producto de esta restricción fue analizado por electroforesis en un gel de agarosa al 2% y observado en un visor de luz ultravioleta para evidenciar y comparar los patrones obtenidos con los patrones estándar del virus de la BIA (Massachusetts, Arkansas, cepas relacionadas con Arkansas, Connecticut, JMK y la 072 ). Como control positivo se utilizó una cepa del serotipo Massachussets la cual fue suministrada por el laboratorio del Poultry Disease and Research Center de la Universidad de Georgia, USA y como control negativo se utilizó un FA libre del virus de la BIA. RESULTADOS Y DISCUSIÓN Extracción del ARN. El ARN fue extraído de los 9 fluidos alantoideos en los cuales se sospechaba la presencia del virus de la BIA por las lesiones macroscópicas observadas en embriones. Las concentraciones de ARN se determinaron por espectrofotometría. Las concentraciones oscilaron entre 150 y 280 ng/ml. RT-PCR. En todas las muestras se amplificó el gen que codifica las proteínas N, M y Sl. En relación con el marcador molecular se observó un peso molecular de un producto de PCR N/M = 1 000 kbp (par de kilo bases nitrogenadas) y de Sl = 1900 kbp (Figura 1). RFLP. Todas las cepas mostraron patrones de restricción, lo cual permitió reunir dos grupos ( A y B), el A perteneciente al serotipo Massachussets identificadas P = 02, P = 03, P = 06, P = 11, P = 12, P = 13 y P = 6246, y el B que corresponde a las cepas P = 6228 y P = 6328 cuyo patrón de RFLP no coincidió con el patrón de las cepas estándar, siendo por lo tanto consideradas como desconocidas (Figura 2), y como "variante genotípica". Los resultados del RFLP evidencian el aislamiento, por primera vez en Venezuela, de un serotipo del virus de la BIA distinto al serotipo Massachussets. El gen amplificado de la subunidad Sl fue moderadamente más pesado que los obtenidos por otros investigadores (Kwon et al., 1993; Wang et al., 1996). Los resultados de la RT -PCR se corresponden con los encontrados por Jackwood et al. (1992), los cuales consideran a esta prueba como específica para el diagnóstico de la BIA, debido a que los productos de amplificación no fueron detectados por electroforesis o por hibridización de sondas cuando el patrón usado en la RT -PCR fue extraído de fluido alantoideo infectado con otros virus causantes de enfermedades respiratorias.
El alto porcentaje de identificación del virus de la BIA en el presente estudio (100%), a través de la RT -PCR, indica la alta sensibilidad y especificidad de esta prueba, lo cual concuerda con lo señalado por otros inves- tigadores (Wang el al., 1999), quienes afirman que la PCR fue capaz de identificar el virus de la BIA de fluidos alantoideos, incluso cuando se inocularon huevos embrionados experimentalmente con Mass-41 y Ark 99 simultáneamente, de igual forma en casos de campo la RT -PCR fue capaz de detectar más de un serotipo de virus de la BIA.
La naturaleza de los coronavirus aviares de poseer una gran diversidad antigénica conlleva a la aparición de múltiples serotipos, variantes que seguirán apareciendo y causando enfermedad, de ahí la importancia de los estudios epidemiológicos de la BIA. La variabilidad genética en el gen que codifica la subunidad Sl de la glicoproteína S posee un mecanismo adaptativo del virus a la presión selectiva inmune, asociado con programas intensivos de vacunación y prácticas equivocadas de manejo, la que ha traido como resultado más de 20 serotipos (Keeler et al., 1998). En consecuencia, la determinación rápida y exacta de los serotipos de las cepas de campo es un factor importante para la prevención de la BIA a través de la aplicación de adecuados programas de vacunación. Los resultados obtenidos en este estudio coinciden con los de Knwo et al. (1993), quienes con el uso de las mismas enzimas lograron agrupar 11 cepas de la BIA en 7 serotipos; las cepas variantes analizadas por RFLP en ese estudio tampoco coincidieron con los patrones de RFLP de las cepa de referencia. Por su parte, Wang et al. (1996), mediante la RT-PCR y el análisis del RFLP, obtuvieron patrones que evidenciaron un nuevo genotipo, el cual era aislado por primera vez en Taiwán, señalando que este método es una vía alternativa para agrupar nuevas cepas de la BIA. Los resultados obtenidos señalan la necesidad de continuar estudios con estas cepas denominadas variantes genotípicas, que permitan la caracterización genómica definitiva, así como conocer la presencia de ésta en otra zonas avícolas del país. SUMMARY A study was perform in order to characterize the genome of nine Avian Infectious Bronchitis virus strains isolated from broiler chickens grown in farms located in the Venezuela central region. The technique used was the Reverse Transcription-Polymerase Chain Reaction-Restriction Fragment Length Polymorphism (RT -PCR-RFLP). From 9 isolated strains, 7 corresponded to Massa-chusetts serotype and 2 corresponded to an unknown serotype which did not match any standard restriction patterns; this serotype was identified as "Genotypic Variant". Massachusetts is the only serotype that had been isolated in Venezuela up to now. The presence of unknown serotypes might arise due to inadequate management practices; hence the importance of molecular identification of field strains with the aim to establishing adequate prevention and control programs. According to obtained results, the RT -PCR-RFLP proved to be highly sensitive and specific for Avian Infectious Bronchitis virus diagnosis. Keys Words: Infectious bronchitis; genotypic variant; serotype; RT -PCR- RFLP. BIBLIOGRAFÍA CRINION, R.A. P., R.A. BALL and M. S. HOFSTAD.1970. Pathogenesis of oviduct lesions in immature chickens fo11owing exposure to infectious bronchitis virus at one day old. Avian Dis. 14:32-41. GELB, J. Jr. and M. W. JACKWOOD. 1998. A laboratory manual for the isolation and identification of avian pathogens. Infectious bronchitis. 4th. ed. Published by The American Association of A vian Pathologists. 169-174. GELB, J. .Jr., J. B WOLFF and C. MORAN. 1991. Variant serotypes óf Infectious Bronchitis Virus isolated from commercial layer and broiler chickens. Avian Dis. 35:82-87. GELB, J. Jr., C. KEELER, W. NIX, J. ROSENBERGER and S. CLOUD. 1997. Antigenic and S-l genomic characterization of the Delaware variant serotype of infectious bronchitis virus. Avian Dis. 41:661-669. JACKWOOD, M. W. 1999. Advanced diagnostic techniques and new developments: Infectious bronchitis virus. Memorias del Mondial Vet. Lyon, France. p. 23-26. JACKWOOD, D. and M. JACKWOOD. 1998. Molecular identification procedures In: A laboratory manual for the isolation and identification of avian pathogens. 4th. ed. AAAP. 267-269. JACKWOOD, M. W, H.M. KWON and D. A. HILT. 1992. Infectious Bronchitis Virus detection in alantoic fluid using the polimerase chain reaction and DNA probe. Avian Dis. 36:403-409. KEELER, C.L.Jr., K.L.REED,W.A.NIX and J.Jr. GELB.1998. Serotype identification of Avian Infectious Bronchitis Virus by RT -PCR of the peplomer (S-l) gene. Avian Dis. 42:275-284. KING, D. J. and D. CAVANAGH.1991. Infectious Bronchitis Virus. In: B. W. Calnek, H, J. Barnes, C. W. Beard, W. M. Reid and H. W. Yoder, Jr.; eds. Diseases of Poultry 9th ed. Iowa State University Press. Ames, Iowa. pp.471-484. KWON, H. M., M. W. JACKWOOD and J. Jr. GELB.1993. Differentiation of Infectious Bronchitis Virus serotypes using polymerase chain reaction and restriction fragment length polymorphism analysis. Avian Dis. 37:194-202. MUNNER, M. A., J. A. NEWMAN, D. A. HALVORSOR, V. SIVA- NANDAN and C. N. COM. 1987. Effect of A vian Infectious Bronchitis Virus (Arkansas strain) on vaccinated laying chickens. Avian Dis. 31:820-828. PICAULT, J. P., P. DROUN, M. GUILTER, G. BENNJEAN, J. PROTAIS, R. LHOSPITALIER, J. P. GILLET, J. LAMANDE and A. L. BACHELIER. 1986. Isolation characterization and preliminary cross protection studies with a new pathogenic avian infectious bronchitis virus. (strain PD- 84084). Avian Pathology 15:367-383. WANG, C. H., M. C. HSIEH and P. C. CHANG. 1996. Isolation, pathogenicity, and H-120 protection efficacy of Infectious Bronchitis Viruses isolated in Taiwan. Avian Dis. 40:620-625. WANG, C. H., B.MIGUEL, W. AUSTIN and R. KEIRS: 1999. Comparison of the immunofluorescent assay and reverse transcription polymerase chain reaction to detect and type Infectious Bronchitis Virus. Avian Dis. 43:590-596. |
||||
|
|