|
Veterinaria Tropical > Colección > Sumario > Volumen 28 > Articulo |
Veterinaria Trop. 28(1): 37-48. 2003 CARACTERIZACIÓN MOLECULAR DE DOS CEPAS DEL VIRUS DE LA ENFERMEDAD INFECCIOSA DE LA BURSA Carmen de Noguera*, Morela de Rolo*, C. T. DÍaz
,*** Antonio Herrera**, *Investigadores y ***Profesora. Recibido: octubre 09, 2003. |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
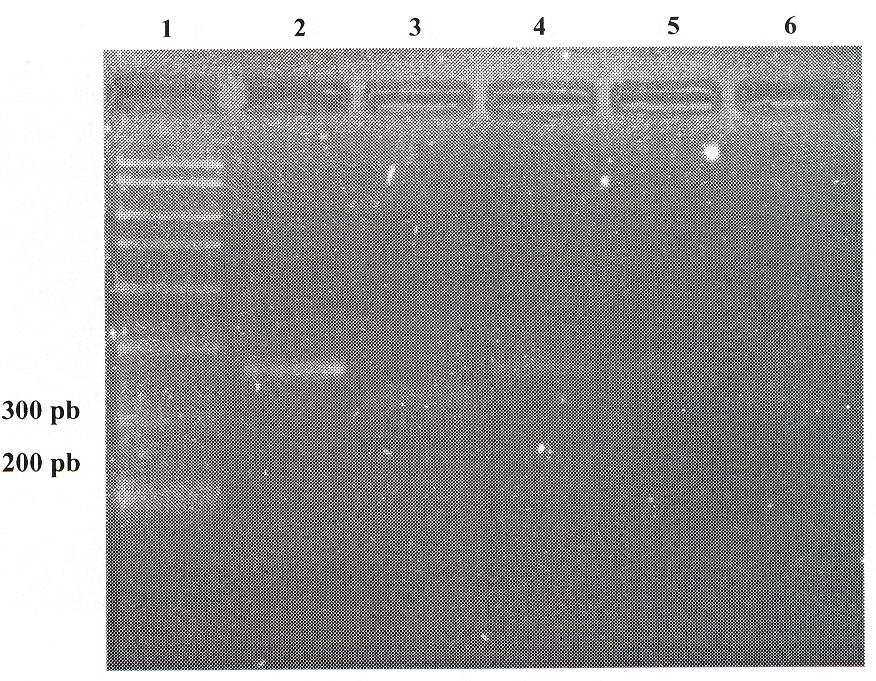
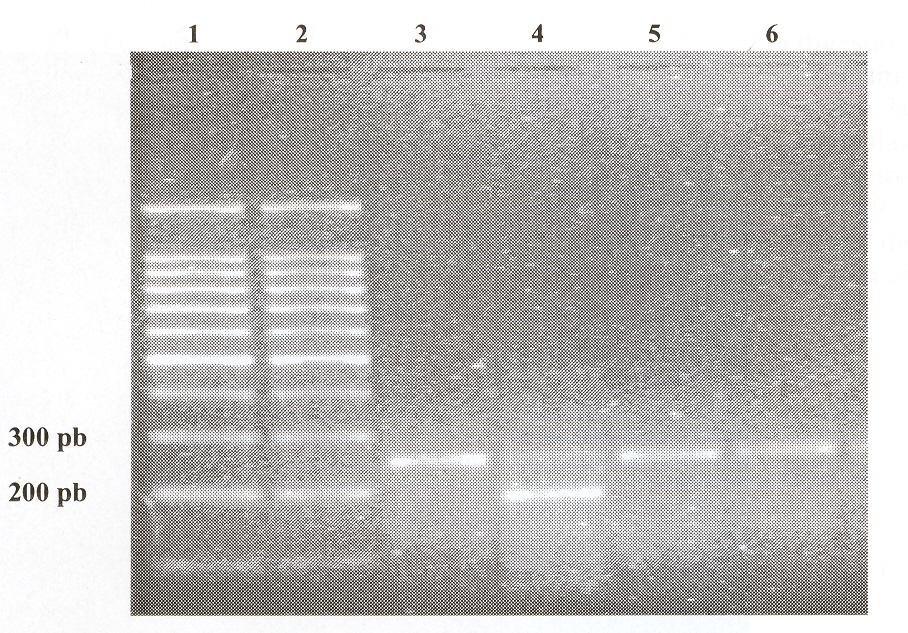
RESUMEN Se caracterizaron dos cepas del virus de la enfermedad infecciosa del bursa (EIB) aislada de pollos de engorde en Venezuela. La técnica de Reacción en Cadena de la Polimerasa (RT-PCR), se utilizó en la transcripción y amplificación de un producto de 248 pares de base que abarca la región hipervariable del gen VP2 (que codifica la proteína) del virus de la EIB. Para el estudio del Polimorfismo en la longitud de los Fragmentos de Restricción (RFLP), el producto de RT-PCR de 248 pb se sometió a la digestión con las enzimas de restricción Dra 1, Sac 1, Sty 1, Ssp I y Taq 1. La cepa P:6228 sólo fue digerida con la enzima Sac I que corresponde con el perfil de las cepas clásicas, mientras el producto de la cepa P:6792 fue digerida con las enzimas Sac I y Taq 1, perfil que identifica las cepas variante E. Ninguna de las cepas mostró la digestión con la enzima Ssp I que identifica las cepas muy virulentas. Palabras Clave: Enfermedad infecciosa del bursa; RT-PCR-RFLP; caracterización molecular. INTRODUCCIÓN La enfennedad infecciosa del bursa (EIB) es una enfennedad aguda altamente contagiosa de los aves jóvenes, siendo las aves de 3 a 6 semanas las más susceptible, aúnque puede afectar aves de menor edad. La enfermedad fue descrita por primera vez por Cosgrave (1962) y es causada por un virusARN segmentado de doble cadena perteneciente a la familia Bimaviridae (Nick et al., 1976). El virion es icosaedrico, desnudo y con un diametro de 58-60 nm (Jackwood et al., 1982). En Venezuela el primer aislamiento del virus de la EIB se realizó a partir de pollos de engorde del oriente del país (Quiroz et al., 1984), identificándose dos serotipos del virus de la EIB I y II (Jackwood y Saif 1987; McFerran et al., 1980), sin embargo, se observan variantes del serotipo I (Jackwood y Jackwood, 1997; Takeshita, 1998; Banda et al., 2001; Ramos et al., 1996; Noguera et al., 2002). El virus del la EIB afecta el sistema inmune de las aves causando inmunosupresión (Allan et al., 1972; Faragher et al., 1974), con las consecuentes pérdidas económicas, en primer lugar por la clínica de la enfermedad y en segundo lugar, la cual es considerada la más importante por la inmunosupresión severa y prolongado que causa en las aves infectadas a temprana edad (Lukert y Saif, 1997), predisponiendo a las mismas a sufrir infecciones bacterianas yvirales, así como fallas en las vacunaciones (Yuasa et al., 1980), por la incapacidad del ave para responder a la aplicación de las vacunas (Quiroz, 1999). A pesar de que el virus de la EIB reduce la capacidad de opsonización de los anticuerpos, este efecto se incrementa marcadamente cuando está presente el virus de bronquitis infecciosa aviar (Naqi et al., 2001), quedando las aves predispuestas a sufrir infecciones del tracto respiratorio como las causadas por E. Coli. Las cepas del virus de la EIB pueden ser identificadas mediante el uso de la prueba de virus-neutralización por la inoculación de huevos embrionados (Jackwood y Saif, 1987; Rosenberger et al., 1998). Igualmente los anticuerpos monoclonales han sido utilizados para conocer las bases moleculares de la variación antigénica de cepas del virus de la EIB (Vakhria et al., 1994) y en la caracterización antigénica y molecular de cepas de campo del virus de la EIB (Sellers et al., 1999). La identificación molecular de cepas del virus de la EIB utilizanda la Transcriptasa Reversa (RT)-Reacción en Cadena de la Polimerasa (PCR) - Polimorfismo en la Longitud de los Fragmentos de Restricción (RFLP), se señala en numerosas investigaciones (Banda et al., 2001; Banda et al., 2003; Jackwood y Jackwood, 1997; Jackwood y Sommer, 1997; Ture et al., 1998; Jackwood y Nie1sen, 1997; Majó et al., 2002; Ikuta et al., 2001). Existen trabajos (Phong et al., 2003) del uso de la técnica de PCR anidada con transcripción reversa e inmuno ensayo asociados a enzimas para la detección de la región hipervariable de la proteína VP2, la cual mostró una especificidad del 100% y la capacidad de detectar bajas concentraciones del virus. Investigaciones recientes, han permitido diferenciar cepas vacuna1es y cepas de campo del virus de la EIB, mediante el uso de la enzima de restricción NgoM IV, siendo considerado como un marcador genético, (Jackwood et al., 2001; Jackwood y Sommer, 2002), ya que la digestión del cDNA con esta enzima se detectó en cepas virulentas de campo, así como en cepas virulentas del virus utilizadas en la elaboración de vacunas, encontrando que el cDNA de las cepas no virulentas no son digeridas con esta enzima. El objetivo de este trabajo es señalar la caracterización molecular de dos cepas del virus del EIB aisladas de pollos de engorde utilizando la técnica de RT-PCR-RFLP. MATERIALES Y MÉTODOS Se utilizó la técnica de RT-PCR-RFLP (Majó et al., 2002) en la caracterización molecular de dos cepas del virus del EIB, a partir de bursas de aves que presentaban signos clínicos y lesiones macroscópicas y microscópicas compatibles con las causada por el virus de EIB, dichas cepas fueron identificadas P=6228 y P=6792. Extracción del RNA viral Pools de 5 bursas de cada una de las muestras se maceraron en un tubo tembroock agregando 3 mI de la solución O completa que contiene guanidina tiocianato 4,23M, citrato de sodio 26,4mM, sarcosina 0,53%, B mercapto etanol 0,0076% yagua tratada con dietil pirocarbonato (DEPC), incubándose en baño de María a 37 °C durante 10 min y a 60 °C por 10 min. Se centrifugaron a 14000 rpm por 3 min, tomando 100 μl del sobrenadante, para mezclar con 400 μl de fenol (pH 4,3), 400 μl de solución D completa, 80 μl de cloroformo alcohol-isoamílico y 40 μl de acetato de sodio. Posteriormente todo fue mezclado en un vortex, mantenido en hielo 15’ y centrifugado a 14000 rpm por 3 mino La capa superior se tomó y puso en un tubo nuevo con 500 μl de Isopropanol, congelándose a -20°C durante 1 h para centifugarse a 14000 rpm por 10 min. Se descartó todo el sobrenadante, restituyendo el pellet en 80 μl de Etanol al 75%, donde se centrifugó a 14000 rpm en 3 min, repitiendo el proceso 2 veces más, luego se descartó el sobrenadante, centrifugándose a 14000 rpm por espacio de 5 mino Fueron agregados 50 μl de dimetil sulfoxido (DMSO) al 90% y las muestras conservadas en congelación a -70°C hasta el momento de montar la técnica de RT-PCR. RT-PCR: Para realizar la técnica se tomó 1,0 μl de la muestra conservada en DMSO, colocándose en el Termociclador a 98 °C/5’ para su desnaturalización. Posteriormente se realizó la técnica de RT-PCR, utilizando una mezcla para la RT que contenía 2 μl de buffer, 0,12 μl de Transcriptasa Reversa M-MLV, 0,22 μl de RNAsin, 1,0 μl de dntp's, 4,66 μl de H20 tratada con dietilpirocarbonato, 1 μl de Primers y la mezcla para el J:>CR contenía 1,8 μl de MgCl2 0,025M; buffer 5 μl; Taq polimerasa 0,2 μl; H20 tratada con dietil pirocarbonato 32 μl y primers 1 μl. En la técnica se usaron dos tipos de primers, el 5’ GTAACAATCACACTGTICTCAGC 3’ utilizado en la mezcla para la RT y e15’ GATGTAACTGGCTGGGTIATCTC 3’ para la mezcla del PCR. A la muestra desnaturalizada se le agregaron 9 μl de la mezcla para RT y 40 μl de la mezcla para PCR, los tubos con la mezla se colocaron en el termociclador para realizar el proceso de transcripción inversa y amplificación, utilizando un termociclador Perkin Elmer Gene Amp PCR System 2400. La transcripción inversa se realizó a 37°C, 1 h. En la amplificación se utilizó el programa siguiente: 1 ciclo a 94°C, 3 min; 40 ciclos a 94°C por 40 segundos, 55°C, 1,5 min; 72 °C, 1 min y 1 ciclo a 72 °C durante 5 mino Con el RT-PCR se amplificó una secuencia de 248 pares de base (Pb) del gen VP2. Al final del proceso se comó una electróforesis para visualizar el producto del RT-PCR, utilizando un gel de agarosa al 2% y coloreado con bromuro de etidio, utilizando como marcador de peso molecular un ladder de 100 pb. Polimorfismo en la longitud de los fragmentos de restricción (RFLP) El producto de 248 pb obtenido del RT-PCR se sometió a la digestión con las enzimas de restricción Ora I , Sac I, Sty I, Ssp I y Taq I. Las enzimas se usaron a una concentración de l0U/μl , por lo tanto la mezcla para la digestión contenía 1 μl de cada una de las enzimas, 2 μl de buffer, 2 μl de la muestra y se completó con H2O para biología molecular hasta obtener un total de 20 μl, las mezclas se dejaron en incubación a 37°C por una noche. Para conocer los resultados de la digestión, se montó una electroforesis utilizando un gel de agarosa al 2% y coloreado con bromuro de etidio y el mismo marcador de peso molecular usados en la RT-PCR. RESULTADOS Y DISCUSIÓN Han sido numerosos los trabajos realizados con la técnica de RT-PCRRFLP en la caracterización de cepas del virus de la EIB utilizando bandas de diferentes tamaños de acuerdo con el número de aminoácidos. Jackwood y Sommer (1998) clasificaron cepas vacunales y de campo del virus de la EIB en cinco grupos moleculares, utilizando un fragmento de 743 pb Y encontrando una alta heterogenicidad entre las cepas. En este estudio la presencia del virus en las muestras procesadas fue confirmado con la formación de una banda entre 200-300 pb observada en la electroforesis del producto obtenido en la transcripción y amplificación. Algunos autores han realizado la caracterización molecular de cepas del virus de la EIB utilizando un producto de tamaño similar al usado en esta investigación (Banda et al., 2001; Bruta et al., 2001; Majó et al., 2002; Banda et al., 2003), mientras que otros investigadores (Jackwood y Jackwood, 1994, 1997; Ture et al., 1998) trabajaron con 394 pb Y 743 pb, respectivamente, por su parte Meir et al. (2001), trabajó con un fragmento de 771 pb, siendo utilizados en todos los casos, fragmentos de diferentes tamaños en la caracterización molecular de cepas del virus del EIB, procedentes tanto de cepas de campo como vacunales. En el Cuadro se presentan los resultados del RT-PCR y de la digestión con las enzimas de restricción (RFLP) del producto obtenido con la amplificación por medio del RT-PCR. Como se observa en el Figura 1, la cepa 6792 fue digerida con las enzimas Sac I y Taq I, resultados similares a los obtenidos en este estudio son presentados en otras publicaciones (Jackwood y Jackwood, 1994, 1997; Jackwood y Nielsen, 1997; Banda et al., 2001), correspondiendo las cepas con este patrón de restricción a las cepas variante E, la cual ya había sido señalada en el país (Ramos et al., 1996; Noguera et al., 2002).
La presencia de esta variante del serotipo 1 es preocupante ya que puede infectar aves jóvenes ocasionando una inmunosupresion prolongada (Lukert y Saif, 1997). En la Figura 2 se observa que el producto RT-PCR de la cepa P:6228, sólo fue digerido con la enzima Sac 1, un patrón similar fue obtenido por Jackwood y Nielsen (1997) cuando usaron las mismas enzimas, clasificando la cepa con este perfil como una cepa desconocida 2, por su parte Jackwood y Jackwood(1994), obtuvieron un patrón similar con la cepa clásica CU-l al utilizar las mismas enzimas usadas en esta investigación. En Brazil, Ikuta et al. (2001) caracterizaron cepas vacunales y de campo originarias de Brazil, encontrando que tres de las cepas vacunales mostraron un patrón similar al obtenido en esta investigación con la cepa P:6228, la cual sólo presentó digestión con la enzima Sac 1, concordando estos resultados con lo señalado para la cepa clásica Cul-D00867. Los resultados obtenido en los estudios realizados a esta cepa, penniten clasificarla como una cepa clásica.
Estudios recientes (Banda et al., 2003) han identificado en Venezuela la presencia de las cepas GLS, Lukert, Edgar y cepas no tipificables. Ninguno de las cepas estudiadas fueron digeridas con la enzima Ssp I, la digestión con esta enzima ha sido observada en cepas de alta virulencia (Banda et al., 2001; Ikuta et al., 2001; Jackwood y Sommer, 1999; Meir et al., 2001), por lo que se considera necesario incorporar esta enzima en las pruebas de RFLP, ya que ello puede ser de gran ayuda en la clasificación de este tipo de cepas. El uso de marcadores epidemiológicos como es el caso de la enzima de restricción NgoM IV, puede ser de gran utilidad en la diferenciación de cepas patógenas de campo y vacunales cuando no se utiliza en laelaboración de las mismas cepas virulentas (Jackwood et al., 2001; Jackwood y Sommer, 2002).
Se puede concluir que el uso de la técnica de RT-PCR-RFLP en la caracterización molecular de cepas del virus de la EIB, es de gran utilidad, ya que ha permitido la diferenciación de cepas del virus (Jackwood y Sornmer, 1997, 1999; Jackwood y Jackwood, 1994; Majó et al., 2002; Banda et al., 2003). Desde el punto de vista epidemiológico es importante conocer el serotipo predominante en una región en particular, ya que ayudatía mucho en el momento de un posible cambio en los biológicos que están siendo utilizados en la prevención y control de la enfermedad. AGRADECIMIENTO Damos las gracias a la Dra. Natalia Majó y al resto del personal de la Universidad Autónoma de Barcelona, España, por su asesoría .en la aplicación de la técnica. Igualmente agradecemos al laboratorio BIOMED por el uso de sus equipos. SUMMARY Two samples of infectious bursal disease (EIB) virus isolated from Venezuelan broilers chickens were characterized. The Reverse Transcriptase - Polymerase Chain Reaction (RT-PCR) technique was used in the transcription and amplification for the 248 base pairs, encompassing the hypervariable region of VP2 gene of the IBD virus. For the study the Restriction Fragment Lenght Polimorphism (RFLP), the product ofthe RT-PCR of248 bp, were digested using the restriction enzymes Dra I, Sac I, Sty I, Ssp I and Taq I. The strain P:6228 was only digested with the enzyme Sac I that corresponds with the profile of the classic strains, while the product ofthe strain P:6792 was digested with the enzymes Sac I and Taq I, profile that identifies the strain variant E. None ofthe strains was digested with the enzyme Ssp 1, which identifies very virulent strains. Key Words: Infectious bursal disease; RT-PCR-RFLP; molecular characterization. BIBLIOGRAFÍA ALLAN, W. H., J. T. FARAGHER and G. A. CULLEN. 1972. Immunosuppression by the infectious bursal agent in chickens immunized againstNewcastle disease. Vet. Rec. 90:511-512. BANDA, A., P. VILLEGAS, J. EL-ATIRACHE and C. ESTEVEZ. 2001.Molecular characterization of seven fields isolates of infectious bursal disease virus obtained from commercial broiler chickens. Avian Dis 45.620-630. BANDA, A., P. VILLEGAS and J. EL-ATTRACHE. 2003. Molecular characterization of infectious bursal disease virus from commercial poultry in The United States and LatinAmerica. Avian Dis 47:87-95. COSGROVE, A. S. 1962. An apparently new disease of Chikens-avian nephrosis. Avian Dis. 6:385-389. FARAGHER, J. T., W. H. ALLAN and C. J. WYETH. 1974. Immunosuppressive effect of infectious bursal agent on vaccination against Newcastle disease. Vet. Rec. 95:385-388. IKUTA, N., J. EL-ATTRACHE, P. VILLEGAS, M. GARCÍA, V. R. LUNGE, A. S .K. FONSECA, C. OLIVEIRA and E. K.MARQUES. 200l. Molecular characterization of Brazilian infectious bursal disease viruses. Avian Dis. 45:297-306. JACKWOOD, D. J. and R. J. JACKWQOD. 1997. Molecular identification of infectious bursal disease virus strains. Avian Dis 41:97-104. JACKWOOD, D. J. and R. J. JACKWOOD. 1994. Infectious bursal viruses: Molecular differentiation of antigenic subtypes among serotipe 1 viruses. Avian Dis. 38:531-537. JACKWOOD, D. J. and C. K. NIELSEN. 1997. Detection ofinfectious bursal disease viruses in commercially reared chikens using The Reverse Transcriptase/Polymerase Chain Reaction-Restriction endonuclease Assay. Avian Dis. 41:137-143. JACKWOOD, D. J. and Y. M. SAIF. 1987. Antigenic diversity of infectious bursal disease viruses. Avian Dis. 31:766-770. JACKWOOD, D. J. and S. E. SOMMER. 2002. Virulent vaccine strains of infectious bursal disease virus not distinguishable from wild-type viruses with the use of a molecular marker. Avian Dis 46:1030-1032. JACKWOOD, D. J. and S. E. SOMMER. 1999. Restriction fragment length polymorphisms in the VP2 gene of infectious bursal disease viruses from outside the United States. Avian Dis 43:310-314. JACKWOOD, D. J. and S. E. SOMMER. 1998. Genetic heterogenicity in the VP2 gene of infectious bursal disease viruses detected in commercially reared chickens. Avian Dis. 42:321-339. JACKWOOD, D. J. and S. E. SOMMER. 1997. Restriction fragment lenght polymorphism in VP2 gene ofthe infectious bursal disease viruses. Avian Dis. 41 :627-637. JACKWOOD, D. J., E. H. BEYERLEY and S. E. SOMMER. 2001. Use of a genetic marker for wild-type potentially pathogenic infectious bursal disease viruses. Avian Dis 45:701-705. JACKWOOD, D. J., Y. M. SAIF and J. H. HUGHES. 1982. Characteristics and serologic studies of two serotypes of infectious bursal disease virus in Turkeys. Avian Dis 26:871-882. LUKERT, P. D. and Y. M. SAlE 1997. Infectious bursal disease. In: Disease of poultry, 10th ed. Edited by B. W. Calnek, H. 1. Bames, C. W. Beard, L. R. McDougald and Y. M. Saif. eds. lowa State University Press, Ames lowa. p. 721-738. MAJÓ, N., J. EL-ATTRACHE, A. BANDA, P. VILLEGAS, A. RAMIS, A. PAGÉS and N. IKUTA. 2002. Molecular characterization ofspanish infectious bursal disease virus field isolates. Avian Dis 46:859-868. McFERRAN,J. B., M. S. McNULTY; E. R. MCKILLOP, T. J.CONNER, R. M. MCCRACKEN, D. S. COLLINS and G. M. ALLAN. 1980. Isolation and serological studies with infectious disease viruses from fowl, Turkey and duck. Demonstration of a second serotype. Avian Pathol. 9:395-404. MEIR, R., D. J. JACKWOOD and Y. WEISMAN. 2001. Molecular typing of infectious bursal disease virus ofIsraeli field and vaccine strains by the Reverse Transcription/Polymerase Chain ReactionlRestriction fragment lenght polymorphism Assay. Avian Dis 45:223-228. NAQI, S., G. THOMPSON, B. BAUMAN and H. MOHAMMED. 2001. The exacerbating effect of infectious bronchitis virus infection on the infectious bursal disease virus induced suppression of opsonization by Escherichia coli antibody in Chickens. Avian Dis. 45:52-62. NICK, H., D. CURSIEFEN and H. BECHT. 1976. Structural and growth characteristics ofinfectious bursal disease virus. J. Virol. 18:227-234. NOGUERA, C. de, M. de ROLO, A. GODO Y, A. HERRERA, J. MARTÍNEZ, L. de ÁVAREZ, M. BRETT. 2002. Agentes infecciosos que afectan la salud aviar. Memorias Proyectos de Investigación, Producción y Servicio. INIA-CENIAP. PBONG, S. E, M. HAIR-BEJO, A. R. OMAR and I. AINI. 2003. Sequence analysis of malasyan infectious bursal di seas e virus isolate and the use of Reverse Transcriptase Nested Polymerase Chain Reaction Enzyme-Linked immunoabsorbent assay for the detection of VP2 hypervariable Region. Avian Dis 47:154-162. QUIROZ, C., C. de NOGUERA, D. INFANTE, A. J. LEÓN, A. BERRERA Y P. VALDILLO. 1984. Primer aislamiento en Venezuela del virus de la enfermedad infecciosa de la bursa (Gumboro). Veterinaria Trop. 9:39-46. QUIROZ, M. A. 1999. Factores que causan inmunosupresión en las aves y su impacto económico. In: Memorias III Seminario Avícola. México. Enfermedades infecciosas inmunosupresoras. p. 12-18. RAMOS, J., R. FERNÁNDEZ, E. MONTIEL, C. JARAMILLO y D. JACKWOOD. 1996. Caracterización de las cepas del virus de la enfermedad de Gumboro en aves de engorde en Venezuela, utilizando la técnica de reacción en cadena de la polimerasa. In: Memorias VI Congreso Nacional de Avicultura. Venezuela. p. 517-523. ROSENBERGER, J. K., Y. M. SAIF and D. J. JACKWOOD. 1998. Infectious bursal disease. A Laboratory manual for the isolation and identification of avian Pathogens 4th ed. D.E. Swayne, Chairman, J. R. Glisson, M. W. Jackwood, J. E. Pearson, Willie M. Reed. 215-218. SELLERS, H. S., P. N. VILLEGAS, B. S. SEAL and D. J. JACKWOOD. 1999. Antigenic and molecular characterization of three infectious bursal disease virus field isolates. Avian Dis 43:198-206. TAKESHITA, K. 1998. Practical use of molecular IBD Identification. Vineland Update N° 62. TURE, O., Y. M. SAIF and D. J. JACKWOOD. 1998. Restriction fragment length polimorphism analysis of highly virulent straints of infectious bursal disease viruses from Holland, Turkey, and Taiwan. Avian Dis. 42:470-479. VAKHARIA, V. N., J. HE, B. AHAMED and D. B. SNYDER. 1994. Molecular basis of antigenic variation in infectious bursal didease virus. Virus Research. 31:00-00. YUASA, N., T. TANIGUCHI, T. NOGUCHI and I. YOSHIDA. 1980. Effect of infectious bursal disease virus infection on incidence of anemia by chicken anemia agent. Avian Dis. 24:202-209. |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||